Wytyczne zostały opracowane przez Margaret J. Hosie, Katrin Hartmann, Reginę Hofmann-Lehmann, Dianę D. Addie, Uwe Truyen, Hermana Egberinka, Séverine Tasker, Tadeusza Frymusa, Marię Grazia Pennisi, Karin Möstl i wsp. oraz zaktualizowane przez Margaret J. Hosie i innych (data aktualizacji 31 stycznia 2021).
Wstęp
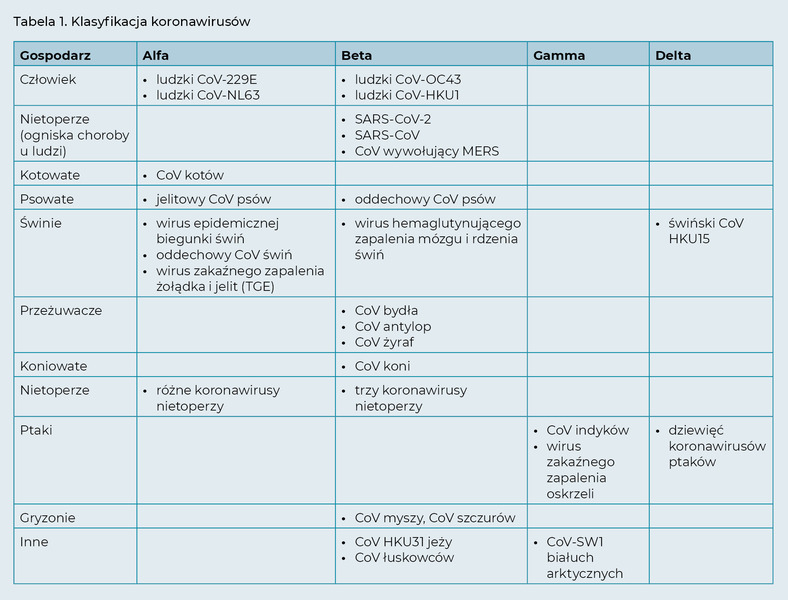
Koronawirus (CoV), który jest przyczyną światowej pandemii COVID-19, został wyizolowany po raz pierwszy w grudniu 2019 r. w mieście Wuhan w prowincji Hubei w Chinach. Nowy wirus jest blisko spokrewniony z koronawirusem zespołu ostrej niewydolności oddechowej (SARS-CoV), który był przyczyną epidemii w 2003 roku. Otrzymał zatem nazwę koronawirus SARS 2 (SARS-CoV-2). SARS-CoV-2 należy do rodzaju Betacoronavirus, rodziny Coronaviridae, rzędu Nidovirales (tab. 1). Jest to nowy szczep, który wcześniej nie był zidentyfikowany u ludzi ani zwierząt. SARS-CoV-2 nie występował u żadnego zwierzęcia towarzyszącego, nie jest też podobny do powszechnie występującego koronawirusa kotów (FCoV) wywołującego zakaźne zapalenie otrzewnej kotów (FIP). Zakażenie SARS-CoV-2 rozprzestrzeniło się u ludzi w wielu krajach na całym świecie, co doprowadziło do ogłoszenia w dniu 11 marca 2020 r. pandemii przez Światową Organizację Zdrowia (WHO, 2020).
Inne koronawirusy ludzi
Do tej pory zidentyfikowano siedem ludzkich koronawirusów (HCoV) (Corman i wsp., 2018; Cui i wsp., 2019), które przedstawiono w tab. 1. Wszystkie mogą powodować choroby układu oddechowego u ludzi, począwszy od bezobjawowego zakażenia lub łagodnego przeziębienia po zapalenie płuc i zapalenie oskrzelików.
W ciągu ostatnich dwudziestu lat u ludzi miały miejsce dwie poważne epidemie związane z koronawirusami – SARS (Drosten i wsp., 2003) i bliskowschodni zespół oddechowy (MERS) (Zaki i wsp., 2012). Zarówno wirusy SARS, jak i MERS wyewoluowały z wirusów krążących u nietoperzy – naturalnego rezerwuaru wielu koronawirusów (Li i wsp., 2005; Ithete i wsp., 2013). Od zwierząt tych wyizolowano wirusy o sekwencjach genetycznych bardzo podobnych do SARS-CoV-2, co wskazuje, że podobnie jak w przypadku poprzednich epidemii nietoperze są prawdopodobnym źródłem nowego koronawirusa. Obecnie nie jest jasne, czy przeniesienie SARS-CoV-2 nastąpiło bezpośrednio z nietoperzy na ludzi, czy też poprzez gospodarza pośredniego.
Trzy z tych siedmiu ludzkich koronawirusów (wywołujące MERS, SARS i COVID-19) mogą powodować poważne choroby, a nawet prowadzić do śmierci, chociaż u niektórych osób zakażenie może być łagodne lub bezobjawowe. Pozostałe cztery ludzkie koronawirusy, które są powszechne w populacji, u zdrowych dorosłych ludzi powodują zwykle jedynie łagodne choroby układu oddechowego. Należy jednak pamiętać, że przyczyniają się nawet do jednej trzeciej przeziębień, a u osób z obniżoną odpornością mogą powodować choroby zagrażające życiu.
SARS-CoV-2 – gatunki zwierząt podatne na zakażenie
Występowanie wirusów u różnych gospodarzy zależy od kilku czynników. Pierwszy etap zakażenia wirusowego związany jest z wiązaniem wirusa z podatną komórką gospodarza w wyniku swoistych interakcji między miejscem wiązania receptora na białku wirusowym a receptorem na komórce gospodarza. Jest to kluczowy czynnik wpływający na wybór gospodarza i tropizm tkankowy wirusa.
Zarówno SARS-CoV, jak i SARS-CoV-2 wykorzystują cząsteczkę enzymu konwertującego angiotensynę 2 (ACE-2) – jednoprzebiegowego białka błonowego typu I – jako receptor umożliwiający wirusowi zakażenie organizmu gospodarza. ACE-2 ulega silnej ekspresji w płucach, tętnicach, sercu, nerkach i jelitach. Jest też ważnym białkiem biorącym udział w regulacji ciśnienia krwi. Oprócz ACE-2, neuropilina-1 ułatwia wnikanie SARS-CoV-2 do komórek i umożliwia jego przechodzenie do ośrodkowego układu nerwowego.
Miejsce wiązania receptora wirusa znajduje się na białku S – glikoproteinie wystającej jak kolec z powierzchni wirusa. Białko S i miejsce wiązania receptora wirusowego SARS-CoV zostały dobrze zbadane, a ostatnio przeprowadzono dodatkowo obszerne analizy sekwencji SARS-CoV-2. Sekwencja ludzkiego SARS-CoV-2 jest podobna do sekwencji koronawirusów krążących u nietoperzy (Zhou i wsp., 2020), które spowodowały epidemię SARS w 2002 r. Podobne wirusy wyizolowano od łuskowców malajskich (Lam i wsp., 2020; Zhang i in., 2020a), co wskazuje, że zwierzęta te mogą być gospodarzami pośrednimi, chociaż nie zostało to jeszcze udowodnione.
Zidentyfikowano trzy krótkie regiony cząsteczki ACE-2 (o długości 3-11 aminokwasów), które są zaangażowane w wiązanie wirusa. Ponadto wykonano analizę porównawczą ich sekwencji u różnych ssaków, w tym ludzi, makaków, koni, świń, kóz, owiec, bydła, kotów, psów, szczurów, myszy, fretek, nietoperzy i cywet. Badania te ujawniły pewne różnice w budowie tych cząsteczek (Li i wsp., 2020). Sekwencje cząsteczki ACE-2 są identyczne u wszystkich małp i ludzi, ale znaleziono różnice u innych gatunków, co może być ważne dla wiązania się wirusa z komórkami. W przypadku kotów i psów jedna część w cząsteczce ACE-2, która jest zasadnicza dla wiązania wirusa, jest inna. Co najciekawsze, sekwencje tej samej cząsteczki u nietoperzy i cywet zawierają dwa istotne fragmenty, które różnią się od sekwencji ludzkiej cząsteczki.
Mimo że zidentyfikowano różnice w budowie receptora ACE-2 u różnych zwierząt, wpływ zmian w sekwencji pojedynczych aminokwasów na jego wiązanie i podatność innych gatunków na zakażenie nie jest jeszcze znany. Wysoka ogólna zbieżność sekwencji mogłaby jednak tłumaczyć stosunkowo szeroki zakres gatunków mogących ulec zakażeniu koronawirusami SARS.
Zakażenia SARS-CoV u kotów
SARS został po raz pierwszy wykryty w 2002 r. na targowisku z owocami morza w Guangdong w Chinach (Drosten i wsp., 2003; Ksiazek i wsp., 2003; Peiris i wsp., 2003). Czynnik sprawczy – SARS-CoV – rozprzestrzenił się z Chin na cały świat (Guan i wsp., 2003; Lee i wsp., 2003; WHO, 2003b), ale po mniej więcej dziewięciu miesiącach epidemia została opanowana dzięki wprowadzeniu ścisłych środków kontroli zakażeń (Guan i wsp., 2003). Zgłoszono 8096 przypadków zachorowań u ludzi i 774 zgony w 27 krajach (WHO, 2003b).
Wykazano, że w warunkach eksperymentalnych SARS-CoV ma zdolność do zakażania wielu gatunków zwierząt, w tym pagum chińskich (łaskunów chińskich) (Wu i wsp., 2005), małp (Fouchier i wsp., 2003; Glass i wsp., 2004; McAuliffe i wsp., 2004; Subbarao i wsp., 2004; Wentworth i wsp., 2004; Roberts i wsp., 2005a; Miyoshi-Akiyama i wsp., 2011), myszy (Roberts i wsp., 2005b; Zaki i wsp., 2012), świń, kur (Weingartl i wsp., 2004), kawii domowych (Liang i wsp., 2005; Mohd i wsp., 2016) oraz chomików syryjskich (Zhao i wsp., 2007; Cui i wsp., 2019). Częstość występowania zakażenia SARS-CoV u pagum chińskich (Paguma larvata) hodowanych do spożycia przez ludzi jest wysoka (Wang i wsp., 2005; Sun i wsp., 2020), a zwierzęta te wytworzyły przeciwciała neutralizujące. Pagumy chińskie uznano za gospodarzy przypadkowych (Kuiken i wsp., 2003; Chen i wsp., 2005), potwierdzono też, że okazjonalnie były bezpośrednim źródłem zakażeń u ludzi (Qin i wsp., 2005; Shi i Hu, 2008; Sun i wsp., 2020).
U kotów potwierdzono możliwość doświadczalnego zakażenia SARS-CoV, które prowadziło do czynnego zakażenia oraz siewstwa wirusa. Ponadto zmiany w płucach były bardzo podobne do tych obserwowanych w śmiertelnych przypadkach notowanych u ludzi (Martina i wsp., 2003). W zakażeniu eksperymentalnym SARS-CoV pobrano od człowieka, który zmarł na SARS, i podano dotchawiczo kotom w średniej dawce 106 jednostek zakaźnych (TCID50). Wirus został wyizolowany od kotów z wymazów pobranych z gardła po 2-8 dniach od zakażenia oraz z nosa w 4. i 6. dniu po zakażeniu. W wymazach z odbytu nie wykryto SARS-CoV. U zakażonych kotów nie zaobserwowano jednak żadnych objawów klinicznych. Cztery dni po zakażeniu cztery zwierzęta poddano eutanazji i wykonano sekcję. SARS-CoV wyizolowano z tchawicy i płuc, potwierdzając zakażenie dolnych dróg oddechowych. Wszystkie koty zakażone SARS-CoV, które nie zostały poddane eutanazji, wytworzyły przeciwciała neutralizujące (miana od 40 do 320 w 28. dniu po zakażeniu). U dwóch kontrolnych kotów, które przebywały razem ze zwierzętami zakażonymi, badaniem RT-PCR także potwierdzono obecność SARS-CoV. Ilość RNA wirusa stopniowo wzrastała u nich od dwóch dni po zakażeniu, osiągając szczyt pomiędzy 6. a 8. dniem. U obu tych kotów nie zaobserwowano żadnych objawów klinicznych, ale w 28. dniu stwierdzono u nich przeciwciała (Martina i wsp., 2003).
W innym badaniu, w którym koty zostały eksperymentalnie zakażone dotchawiczo 106 TCID50 SARS-CoV, również opisano występowanie zmian w płucach (van den Brand i wsp., 2008). W badaniu histologicznym SARS-CoV wykryto w komórkach wykazujących ekspresję receptora ACE-2, a u kotów doszło do rozlanego uszkodzenia pęcherzyków płucnych i wielu innych zmian patologicznych, obserwowanych również u ludzi chorych na SARS (van den Brand i wsp., 2008). Różnica polegała na tym, że u kotów pojawiło się również zapalenie tchawicy i oskrzeli, którego nie odnotowano u ludzi (van den Brand i wsp., 2008, 2014). Dane z dwóch niezależnych badań wykazały, że koty domowe są wrażliwe na eksperymentalne zakażenie SARS-CoV, wirus może być przenoszony na inne koty, a objawy kliniczne i zachodzące procesy patologiczne są podobne do tych występujących u ludzi (Martina i wsp., 2003; van den Brand i wsp., 2008).
Podczas pierwszego wybuchu epidemii SARS opisano również występowanie naturalnego zakażenia u kotów wychodzących. Zwierzęta SARS-CoV-dodatnie zidentyfikowano za pomocą badania RT-PCR (Kuiken i wsp., 2003; Rowe i wsp., 2004; Wang i wsp., 2005; Shi i Hu, 2008; He i wsp., 2013). Ponadto koty domowe utrzymywane w bloku mieszkalnym w Hongkongu, gdzie 100 osób było zakażonych SARS-CoV, także miały dodatni wynik w badaniu RT-PCR. Nie znaleziono jednak ostatecznego dowodu na bezpośrednie przenoszenie wirusa z ludzi na koty (Martina i wsp., 2003; WHO, 2003a; Lawler i wsp., 2006). W ciągu 14 dni po rozpoznaniu SARS u właścicieli zwierząt pobrano wymazy z jamy ustnej i odbytu od kotów z gospodarstw domowych, gdzie mieszkało więcej niż jedno zwierzę, oraz od dwóch psów. W tym badaniu osiem kotów i jeden z psów miały dodatni wynik w badaniu RT-PCR. Również w ciągu 14 dni wykazano za pomocą RT-PCR spontaniczne zakażenie kotów z innych trzech gospodarstw domowych utrzymujących kilka zwierząt (WHO, 2003a).
Od kotów wyizolowano też SARS-CoV. Wirus był nie do odróżnienia od izolatów ludzkich (WHO, 2003a). Testy seroneutralizacji potwierdziły zakażenie SARS-CoV u jednego kota z dodatnim wynikiem RT-PCR z jednego gospodarstwa domowego i u czterech spośród pięciu kotów (w tym trzech kotów z dodatnim wynikiem testu RT-PCR) z innego gospodarstwa. Koty odizolowano i trzymano w oddzielnych pomieszczeniach, przy czym zwierzęta pochodzące z jednego gospodarstwa domowego zamykano w odrębnych klatkach. Stwierdzono ograniczone rozprzestrzenianie się wirusa między izolowanymi klatkami (pięć kotów pozostających w bliskim kontakcie bezpośrednim z tymi zwierzętami pozostało niezakażonych) (Martina i wsp., 2003; WHO, 2003a; Lawler i wsp., 2006).